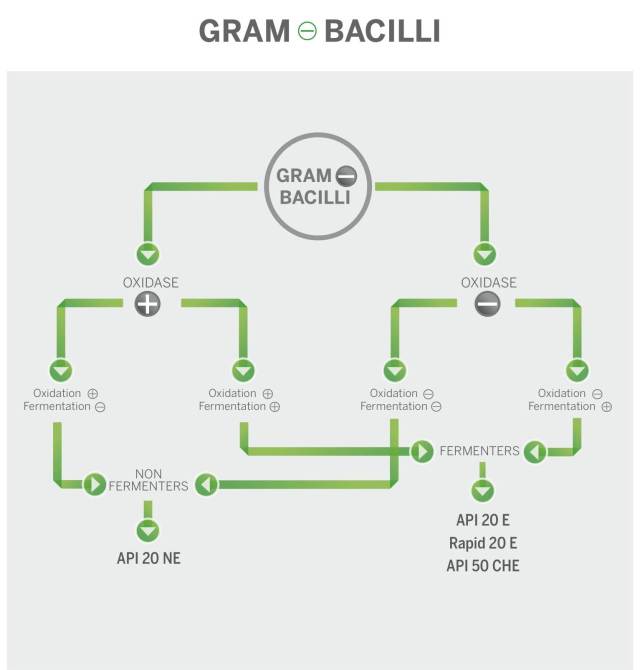
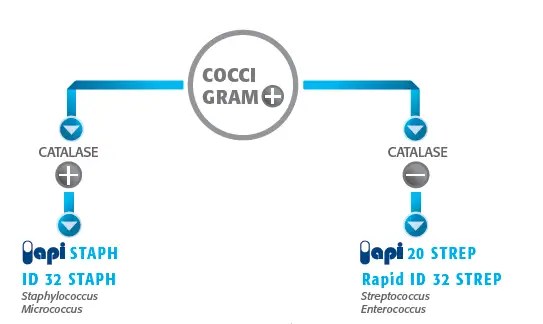
BioMérieux’s API (Analytical Profile Index) identification products are test kits for the identification of Gram-positive bacteria, Gram-negative bacteria, and yeast. The kits include strips that contain up to 20 miniature biochemical tests with a long shelf life.
It is one of the well-established methods for the identification of microorganisms up to the species level. The company runs APIWEB, where laboratories can input numerical profiles of the API strip results to obtain the organism identification. The software database enables reliable automated interpretation of API strip results which can be read on the screen and printed as per the need.
Table of Contents
Identification System and Species Identified
| Identification System | Species Identification |
| API 20 E | Gram-negative bacilli |
| API 10 S | Gram-negative bacilli |
| Rapid 20E | Enterobacteriaceae |
| API 20 NE | Gram-negative non-Enterobacteriaceae |
| API Staph | Staphylococci |
| API 20 Strep | Streptococci |
| API Candida | Yeasts |
| API 20 C AUX | Yeasts |
| API 20 A | Anaerobes |
| API Coryne | Corynebacteria |
| API Campy | Campylobacter |
| API Listeria | Listeria |
| API NH | Neisseria, Haemophilus |
| API 50 CHE | Enterobacteriaceae |
| API 50 CHL | Lactic bacteria |
| API 50 CHB | Bacillus |
| ID 32 E | Gram-negative bacilli |
| Rapid ID 32 E | Enterobacteriaceae |
| ID 32 GN | Gram-negative bacilli |
| ID 32 STAPH | Staphylococci |
| Rapid ID 32 STREP | Streptococci |
| ID 32 C | Yeasts |
| Rapid ID 32 A | Anaerobes |
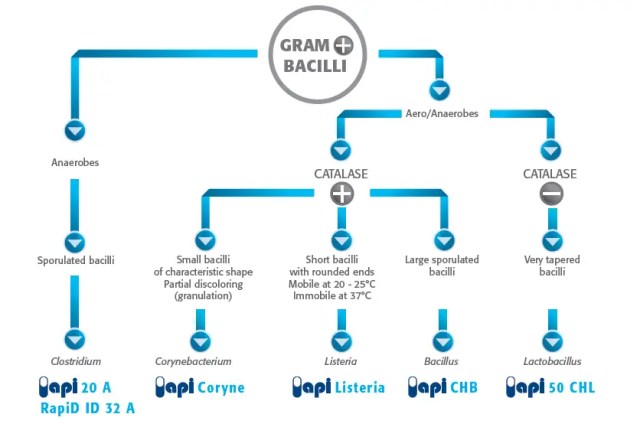
API 20 A
The API 20 A system which consists of 20 microtubules containing dehydrated substrates. It enables 21 tests to be carried out quickly and easily for the biochemical identification of anaerobes. Other tests such as colonial and microscopic morphology, Gram stain, etc. should be performed. The results are used to confirm or complete the identification.
These microtubes are inoculated with a bacterial suspension which reconstitutes the media. During incubation, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents. The reactions are read according to the reading table. The identification is obtained by referring to the analytical profile index (API) or using the identification software.
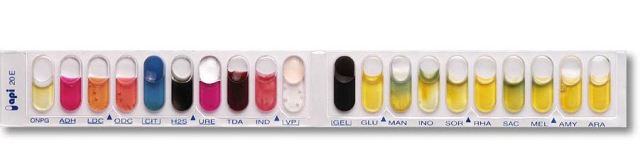
API 20 E
API 20 E is a standardized identification system for Enterobacteriaceae and other non-fastidious, Gram-negative rods which uses 21 miniaturized biochemical tests and a database.
The API 20 E strip consists of 20 microtubes containing dehydrated substrates. These tests are inoculated with a bacterial suspension that reconstitutes the media. During incubation, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents. The reactions are read according to the reading table. The identification is obtained by referring to the analytical profile index (api) or using the identification software.
Find detailed information about API 20 E in this post: API 20E Test System: Results and Interpretations
API 20 NE
API 20 NE is a standardized system for the identification of non-fastidious, non-enteric Gram-negative rods (e.g. Pseudomonas, Acinetobacter, Flavobacterium, Moraxella, Vibrio, Aeromonas, etc.) and a database. The API 20 NE strip consists of 20 microtubes (for 8 conventional tests and 12 assimilation tests) containing dehydrated substrates.
- The conventional tests are inoculated with a saline bacterial suspension which reconstitutes the media. During incubation, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents.
- The assimilation tests are inoculated with a minimal medium. The bacteria grow if they are capable of utilizing the corresponding substrate.
The reactions are read according to the reading table. Then, the identification is obtained by referring to the analytical profile index (API) or using the identification software.
API 50 CHB/ CHE
API 50 CHB/E medium is used for the identification of Bacillus and related genera, as well as Gram-negative rods belonging to the Enterobacteriaceae and Vibrionaceae families. It is a ready-to-use medium that allows the fermentation of the 49 carbohydrates on the API 50 CH strip.
A suspension is made in the medium with the microorganism to be tested and each tube of the strip is then inoculated with the suspension. During incubation, fermentation of carbohydrates is detected by the color change of the pH indicator. The results make up the biochemical profile which is used by the identification software to identify the strain. The API 20 E strip may be used in association with the API 50 CH strip to provide supplementary tests for Enterobacteriaceae and Vibrionaceae.
API STAPH and ID 32 STAPH
API Staph is a miniaturized biochemical test system for the identification of the genera Staphylococcus and Micrococcus.
The API Staph strip consists of 20 microtubes containing dehydrated substrates. These microtubes are inoculated with a bacterial suspension, prepared in API Staph medium, that reconstitutes the tests. During incubation, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents. The reactions are read according to the reading table and the organism is identified by referring to the analytical profile index or using the identification software.
API 20 STREP and Rapid ID 32 STREP
API 20 Strep is a standardized system combining 20 biochemical tests for the identification of most streptococci and enterococci. The API 20 Strep strip consists of 20 microtubes containing dehydrated substrates for the demonstration of enzymatic activity or the fermentation of sugars.

- The enzymatic tests are inoculated with a dense suspension of organisms, made from a pure culture. This then used to reconstitute the enzymatic substrates. During incubation, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents.
- The fermentation tests are inoculated with an enriched medium that rehydrates the sugar substrates. Fermentation of carbohydrates is detected by a shift in the pH indicator.
The reactions are read according to the reading table and the identification is obtained by referring to the analytical profile index or using the identification software.
API (Analytical Profile Index) 20C AUX and ID 32 C
API 20 C AUX and ID 32 C is used for the identification of Candida, Cryptococcus, Geotrichum, Kloeckera, Pichia, Rhodoturela and Trichosporon.
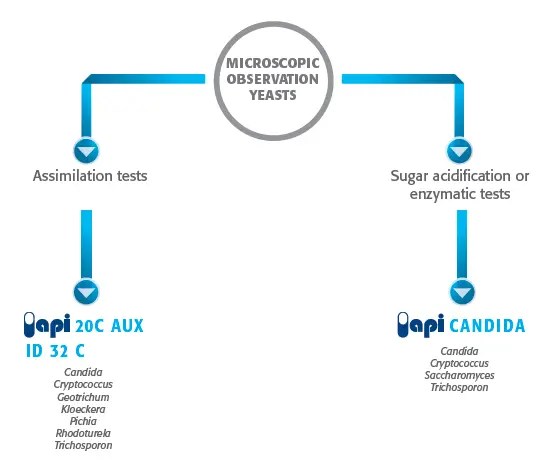
API CANDIDA
API Candida is a standardized system for the identification of frequently encountered yeasts (Candida, Cryptococcus, Saccharomyces, and Trichosporon) in clinical microbiology. The API Candida strip consists of 10 tubes containing dehydrated substrates which enable testing of 12 sugar acidification or enzymatic reactions. The reactions produced during incubation are revealed by spontaneous color changes.
API (Analytical Profile Index) CAMPY
API Campy is a standardized miniature test system for the identification of Campylobacter. The API Campy strip consists of 20 microtubes containing dehydrated substrates. It is made up of two parts.
- The first part of the strip (enzymatic and conventional tests) is inoculated with a dense suspension that rehydrates the substrates. During incubation in aerobic conditions, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents.
- The second part of the strip (assimilation or inhibition tests) is inoculated with a minimal medium and incubated in microaerophilic conditions. The bacteria grow if they are capable of utilizing the corresponding substrate or if they are resistant to the antibiotic tested. The reactions are read according to the reading table.
The organism is identified using the identification profile list in the package insert or by using identification software.
API (Analytical Profile Index) CHL
API 50 CHL is a ready-to-use medium that allows fermentation of 49 carbohydrates and is used for the identification of the genus Lactobacillus and related genera.
A suspension is made in the medium with the microorganism to be tested and each tube of the strip is then inoculated with the suspension. During incubation, the carbohydrates are fermented to acids which produce a decrease in the pH, detected by the change in color of the indicator. The results make up the biochemical profile which is used by the identification software to identify the strain.
API (Analytical Profile Index) Coryne
API Coryne is a standardized miniature test system for the identification of Corynebacterium in 24 hours. It consists of 20 microtubes containing dehydrated substrates for the demonstration of enzymatic activity or the fermentation of carbohydrates.
- The enzymatic tests are inoculated with a dense suspension of organisms, which reconstitutes the enzymatic substrates. During incubation, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents.
- The fermentation tests are inoculated with an enriched medium (containing a pH indicator) which reconstitutes the sugar substrates. Fermentation of carbohydrates results in acidification which is detected by a spontaneous color change in the pH indicator.
The reactions are read according to the reading table and the identification is obtained by referring to the analytical profile index (API) or using the identification software.
API Listeria
API Listeria is a standardized miniature test system for the identification of Listeria. The API Listeria strip consists of 10 microtubes containing dehydrated substrates which enable the performance of enzymatic tests or sugar fermentations.
During incubation, metabolism produces color changes that are either spontaneous or revealed by the addition of reagents. The reactions are read according to the reading table and the identification is obtained by consulting the profile list in the package insert or using the identification software.
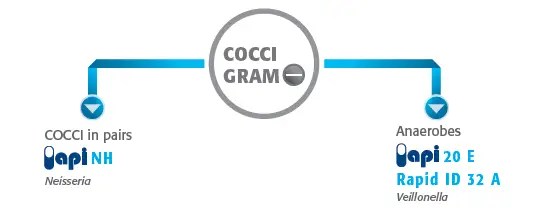
API NH
API NH is a standardized miniature system for the identification of Neisseria, Haemophilus (and related general and Moraxella catarrhalis (Branhamella catarrhalis) using a specially adapted database.
The API NH strip consists of 10 microtubes containing dehydrated substrates, which enable the performance of 12 identification tests (enzymatic reactions or sugar fermentation), as well as the detection of a penicillinase. API NH also enables the biotyping of Haemophilus influenzae and Haemophilus parainfluenzae.
For more information please visit BioMérieux’s API identification
Производитель может изменять цены — запросите коммерческое предложение сейчас, и мы зафиксируем за вами текущую цену.
В случае наличия продукции на складе:
Доставим по Москве за 7 дней,
по России до 30 дней.
Доступен самовывоз
со склада
Получите коммерческое предложение
Менеджер подготовит коммерческое предложение и позвонит, если понадобится уточнить детали вашего заказа

Наш опыт работы в научных институтах и сфере продаж позволяет нам в кратчайшие сроки дать рекомендацию как по выбору расходных материалов, реагентики, так и по подбору необходимого оборудования для ваших исследований
Обрудование, расходные материалы, реагенты, вам подберут сотрудники с высшим химическим образованием
Работаем напрямую с заводами-изготовителями
Быстрая доставка по Москве, а также по России
Храним реактивы по требованиям производителя
Получите коммерческое предложение
Менеджер подготовит коммерческое предложение и позвонит, если понадобится уточнить детали вашего заказа
API 20 E®
Идентификация грамотрицательных палочек за 18-24 часа
• База данных включает практически все встречающиеся в клинической практике виды
• Референсный метод для оценки характеристик других систем идентификации (600 публикаций)
• Быстрота и простота в использовании: нужна одна колония, инструкции по заполнению стрипа указаны прямо на стрипе, стрипы запечатаны в пакеты, цвета реакций четко различимы, специальное программное обеспечение для интерпретации результатов
• Длительный срок хранения
25 стрипов
RapiD 20 E®
Экспресс-идентификация энтеробактерий за 4 часа
• База данных включает практически все встречающиеся в клинической практике виды
• Система адаптирована для критических ситуаций: быстрое назначение терапии, упрощенный режим работы лаборатории
• Достоверный результат: используется стандартизованная суспензия низкой плотности (0.5 единиц МакФарланда), что позволяет избежать пересева и смешанных культур
• Простота в использовании
• Длительный срок хранения
25 стрипов
API 20 NE
Идентификация неферментирующих грамотрицательных палочек за 24-48 часов
• Система выбора для идентификации бактерий-оппортунистов, являющихся частыми возбудителями внутрибольничных инфекций (поскольку эти бактерии становятся все более и более устойчивыми к антибиотикам, точная идентификация необходима)
• Тесты для определения ферментативной активности и способности утилизировать углеводы, адаптированные для неферментирующих бактерий; референсная система в идентификации неферментирующих бактерий
• Достоверный результат: используется стандартизованная суспензия низкой плотности (0.5 единиц МакФарланда), что позволяет избежать пересева и смешанных культур.
25 стрипов
API 10
Идентификация грамотрицательных палочек по упрощенной схеме за 18-24 часа
• Упрощенный вариант стрипа API 20 E (10 тестов вместо 20)
• Высокая надежность
• Быстрота и простота в использовании: нужна одна колония, инструкции по заполнению стрипа указаны прямо на стрипе, стрипы запечатаны в пакеты, цвета реакций четко различимы, специальное программное обеспечение для интерпретации результатов
50 стрипов
API Candida
Идентификация дрожжей, наиболее часто встречающихся в клинической практике, за 18-24 часа
• Эффективность: 12 биохимических тестов для идентификации дрожжей
• Система адаптирована к современным потребностям: идентификация 14 наиболее часто встречающихся в клинической практике видов дрожжей
• Простота в использовании: спонтанное изменение цвета реакции, цвета четко различимы, интерпретация при помощи короткого списка профилей
• Готовый к использованию продукт, не нужно дополнительных реактивов (среды для приготовления суспензии включены в набор)
10 стрипов
API 20 C AUX
Идентификация дрожжей за 18-24 часа
• Тесты для определения способности утилизировать углеводы = референсные тесты в идентификации дрожжей
• Простота в использовании: наличие положительного и отрицательного контроля для облегчения учета результата, не нужно дополнительных реактивов
• Расширенная база данных, включающая практически все виды, встречающиеся в клинической и ветеринарной практике
25 стрипов
API Staph
Идентификация стафилококков и микрококков за 18-24 часа
• Стандартные биохимические тесты и ферментационные тесты = референсные тесты в идентификации стафилококков
• Достоверный результат: используется стандартизованная суспензия низкой плотности (0.5 единиц МакФарланда), что позволяет избежать пересева и смешанных культур
• Очень простой метод: приготовление суспензии осуществляется прямо в питательной среде API Staph
25 стрипов
API 20 Strep
Идентификация стрептококков и родственных бактерий за 4 или 24 часа
• Точная идентификация до вида (что важно для эпидемиологии и антибиотикотерапии, поскольку патогенный потенциал и другие свойства различных видов могут сильно варьировать в пределах одной серологической группы)
• Простота в использовании и достоверный результат: идентификация родственных бактерий, которые иногда трудно определить в окрашенном по Граму мазке и дифференцировать от стрептококков.
25 стрипов
API 20 A®
Идентификация анаэробных бактерий за 24-48 часов
• Ферментационные тесты = референсные тесты в идентификации анаэробов
• Простота в использовании: приготовление суспензии осуществляется прямо в питательной среде API 20 A
• Многопараметрическая система для идентификации всех анаэробных бактерий, грамположительных и грамотрицательных
25 стрипов
API Coryne
Идентификация коринеформных бактерий за 24 часа
• Система выбора для идентификации бактерий-оппортунистов, являющихся частыми возбудителями внутрибольничных инфекций
• Единственная система для идентификации коринебактерий
• Простая, достоверная идентификация коринеформных бактерий, которых трудно охарактеризовать в мазке, окрашенном по Граму
• Стандартные биохимические тесты и ферментационные тесты = референсные тесты в идентификации коринебактерий
• Упаковка содержит 12 тестов, что удобно, поскольку коринебактерии не часто встречаются в клинической практике
12 тестов
API Listeria
Идентификация всех видов рода Listeria за 24 часа
• Единственная система для идентификации всех видов рода Listeria, в том числе Listeria monocytogenes, без CAMP-теста, с использованием оригинального запатентованного биохимического теста (DIM)
• Достоверный результат: в стрип включены специфические тесты для дифференциации рода Listeria от других родов со сходной морфологией; используется стандартизованная суспензия низкой плотности (0.5 единиц МакФарланда), что позволяет избежать пересева и смешанных культур
• Простота в использовании: приготовление суспензии осуществляется прямо в питательной среде
10 стрипов
API NH
Идентификация Neisseria spp., Haemophilus spp. и Moraxellacatarrhalis (Branhamella catarrhalis) за 2 часа
• Одновременная идентификация (Neisseria spp.,Haemophilus spp. и Moraxella catarrhalis), биотипирование (H. influenzae, H. parainfluenzae) и определение пенициллиназы
• Быстрое получение результата (2 часа)
• Простота в использовании
• Упаковка содержит 10 тестов, что удобно, поскольку эти бактерии не часто встречаются в клинической практике
10 стрипов
API Campy
Идентификация Campylobacter spp. за 24 часа
• Единственная система для идентификации кампилобактерий
• Достоверный результат: стандартизованный метод, сочетающий референсные тесты и оптимальные условия для роста бактерий этой группы
• Простота в использовании и эффективность: один этап для идентификации и прогноза чувствительности к антибиотикам (эритромицин)
• Упаковка содержит 12 тестов, что удобно, поскольку кампилобактерии не часто встречаются в клинической практике
12 стрипов
API 50 CH
Система для научно-исследовательской работы (изучение углеводного метаболизма)
Данный стрип состоит из 49 углеводных субстратов и предназначен для:
• Идентификации бактерий рода Bacillus в сочетании со средой API 50 CHB, приблизительно за 48 часов
• Идентификации бактерий рода Lactobacillus в сочетании со средой API 50 CHL за 48 часов
• Идентификации и биотипирования Enterobacteriaceae в сочетании со средой API 50 CHE за 48 часов
• Других научно-исследовательских применений, требующих изучения метаболизма углеводов
10 стрипов
Free access supported by contributions and sponsoring — share your knowledge or support us financially
The API 50 CHL test kits are a standardized identification system for the biochemical characterization of microorganisms. The kits provide a standardized and reproducible method for the identification of Gram-positive and Gram-negative bacteria, based on the observation of bacterial growth and carbohydrate fermentation in a series of miniaturized biochemical tests.
Automatically generated — may contain errors
4 protocols using api 50 chl test kit
1
Identification of Metal-Resistant Lactic Acid Bacteria
Check if the same lab product or an alternative is used in the 5 most similar protocols
Potential metal-resistant LAB isolates were biochemically characterized by API 50 CHL test kits (BioMérieux, Marcy I´Etoile, France) according to the manufacturer’s instructions and interpretation of results was performed using the computer-aided database API-WEB™V.5.0 software as mentioned by Khalil et al.43 . Furthermore, selected isolates were identified by 16S rRNA gene sequencing, following Hamdan et al.44 (link), analyzed in the Genbank DNA database using the online tool (BLAST) at NCBI (
http://www.ncbi.nlm.nih.gov/BLAST/
), and deposited in the DNA Data Base of Japan (DDBJ) for serial accession numbers.
Ameen F.A., Hamdan A.M, & El-Naggar M.Y. (2020). Assessment of the heavy metal bioremediation efficiency of the novel marine lactic acid bacterium, Lactobacillus plantarum MF042018. Scientific Reports, 10, 314.
+ Expand
2
Isolation and Identification of Lactobacillus Strains from Algerian Poultry
Check if the same lab product or an alternative is used in the 5 most similar protocols
The lactobacilli strains were isolated from gizzard contents of Algerian local poultry. Decimal dilution of these samples were mixed with MRS medium and incubated at 37°C for 48 h under anaerobiosis (7 ). Selected colonies were picked from the higher dilutions and sub-cultured in MRS broth. The identification of the isolates was performed according to the criteria of Bergey’s Manual of Determinative Bacteriology and using the methods and criteria of Sharpe (8 ). The isolates were initially subjected to Gram staining and catalase test (3% H2O2). Only the Gram positive, catalase negative isolates were further identified. Growth at different temperatures was determined in MRS broths (10°C, 15°C, 40°C and 45°C). Hydrolysis of arginine was also recorded. The fermentative type was determined on agar.
The ability of the isolated strains to produce acid from different carbohydrates was determined by API 50 CHL test kits (BioMerieux, S.A., France). The results were loaded onto the API system software, which used the phenotypic data to predict a species identity (%) for each isolate.
, & Idoui T. (2014). Probiotic properties of Lactobacillus strains isolated from gizzard of local poultry. Iranian Journal of Microbiology, 6(2), 120-126.
+ Expand
3
Pediococcus Strain Identification Using 16S rDNA and Genomic Sequencing
Check if the same lab product or an alternative is used in the 5 most similar protocols
The species identification of the Pediococcus strain from the 16S rDNA was conducted using an API 50 CHL test kit (bioMérieux, Marcy l´Etoile, France) according to the
manufacturer’s instructions. Next, we used whole-genomic sequence analysis, which was carried out by Takara Bio Inc. (Shiga, Japan). Briefly, the sequence was analyzed by using a PacBio
RS II (Pacific Biosciences, Menlo Park, CA, USA), and the obtained reads were assembled by a genome assemble algorithm, SMRT Analysis v2.3.0 (Pacific Biosciences, Menlo
Park, CA, USA). The obtained contig sequences including the 16S rRNA gene sequence were further analyzed by NCBI BLAST (
https://blast.ncbi.nlm.nih.gov/Blast.cgi
).
FUJIWARA M., KUWAHARA D., HAYASHI M., ZENDO T., SATO M., NAKAYAMA J, & SONOMOTO K. (2020). Lowering effect of viable Pediococcus pentosaceus QU 19 on the rise in postprandial glucose. Bioscience of Microbiota, Food and Health, 39(2), 57-64.
+ Expand
4
Bacterial Catalase and Carbohydrate Utilization
Check if the same lab product or an alternative is used in the 5 most similar protocols
The presence of catalase from a bacterial isolate is obvious when a small amount of bacterial culture from their agar selective medium developed bubbles (effervescence) at addition of 3% hydrogen peroxide (3%, v/v) (MacFaddin, 2000) .
The API 50 CHL All strains were identified (genus and species) and characterized based on biochemical evaluation tests using a bioMerieux API 50 CHL test kit according to the manufacturer’s instructions (BioMerieux, Marcy l’Etoile, France). The test API 50 CHL put in evidence the enzymatic equipment bacteria, which differs from one species to another. The API 50 CHL consists of 50 microtubes used to study carbohydrates fermentation. The fermentation tests are inoculated with bacterial strain suspension in API 50 CHL medium which rehydrates the substrates. During incubation, the ability of isolate to ferment carbohydrates is indicated by the colour change of the basal medium used, caused by the anaerobic production of acid and detected by the pH indicator present in the selective medium. The strips are visually examined after 24-72 h. A positive or negative result was determined from the colour change from purple to yellow. The results were then introduced to the API databases using APIwebTM 50 CHL V5.1 and ABIS online software (Stoica and Sorescu, 2017) .
Dumitru M., Ciurescu G., & Hăbeanu M. (2021). Evaluation ofLactobacillusspp. Based on Phenotypical Profile as Direct-Fed Microbial Candidate for Poultry Nutrition. Archiva zootechnica, 24(2), 150-166.
+ Expand
About PubCompare
Our mission is to provide scientists with the largest repository of trustworthy protocols and intelligent analytical tools, thereby offering them extensive information to design robust protocols aimed at minimizing the risk of failures.
We believe that the most crucial aspect is to grant scientists access to a wide range of reliable sources and new useful tools that surpass human capabilities.
However, we trust in allowing scientists to determine how to construct their own protocols based on this information, as they are the experts in their field.
Ready to
get started?
Sign up for free.
Registration takes 20 seconds.
Available from any computer
No download required
Sign up now
Revolutionizing how scientists
search and build protocols!